Uma equipe de pesquisadores liderada pelo Dr. Sebastian Hess do Instituto de Zoologia da Universidade de Colônia estudou a expressão de enzimas ativas de carboidratos no organismo unicelular Orciraptor agilis por sequenciamento de RNA.
Pela Universidade de Colônia com informações de Phys.
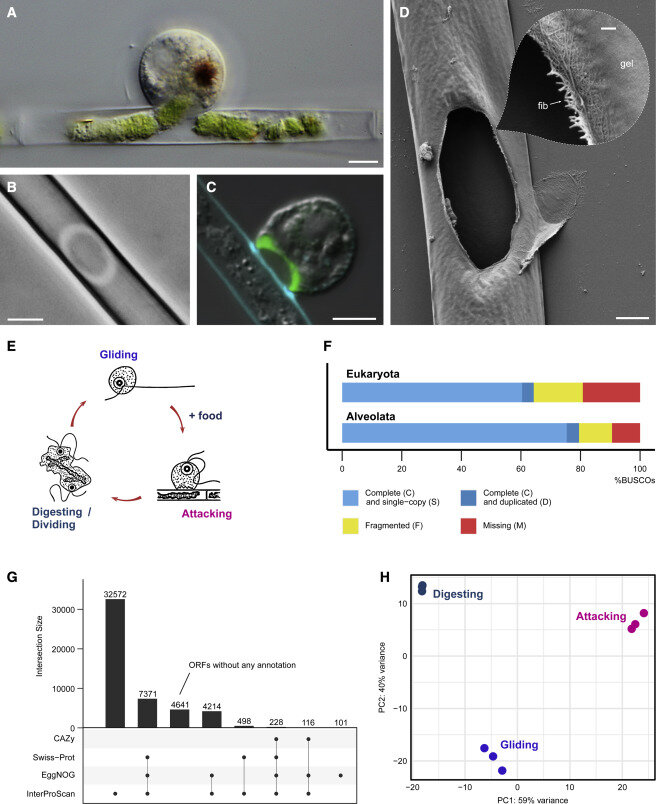
Orciraptor é um chamado “alimentador de protoplastos” e vive exclusivamente do conteúdo celular de algas mortas. Para fazer isso, ele tem que penetrar na parede celular celulósica da presa. Em colaboração com colegas do Centro de Genômica Comparativa e Bioinformática Evolutiva (CGEB) em Halifax, Canadá, os pesquisadores da UoC conseguiram identificar uma possível enzima chave para o ato alimentar altamente especializado do protista.
Ao entrar em contato com as células das algas, o Orciraptor regula positivamente uma enzima que deve ser capaz de clivar a celulose vegetal, com base em sua sequência gênica e estrutura 3D prevista. Esta enzima pode ajudar o protista a dissolver as paredes celulares das algas. Até agora, a base molecular de como os alimentadores de protoplastos interagem com suas presas não era completamente clara. O artigo “Transcritômica comparativa revela o kit de ferramentas molecular usado por um protista algívoro para perfuração da parede celular” na revista Current Biology agora lança alguma luz sobre esse fenômeno.
Além disso, o Orciraptor contém várias proteínas inesperadas, como proteínas de ligação à quitina, uma quitina sintase (Chitin Synthase) e várias quitinases. A função potencial da quitina ou biopolímeros semelhantes no flagelado nu ainda não está clara. No entanto, as enzimas sugerem um importante papel fisiológico da quitina na história de vida do Orciraptor. Enzimas que decompõem biopolímeros recalcitrantes como celulose e quitina também são de grande importância tecnológica e industrial. Atualmente, as aplicações industriais utilizam principalmente enzimas de bactérias e fungos – os organismos tradicionais em biotecnologia microbiana. No artigo publicado, o Dr. Hess e colegas apontam para o potencial biotecnológico ainda inexplorado de microeucariotos não fúngicos como o Orciraptor.
Orciraptor foi descoberto cerca de dez anos atrás em charnecas pobres em nutrientes e descrito pelo Dr. Hess durante seus estudos de doutorado no Instituto de Botânica da Universidade de Colônia. No entanto, existem muitos outros organismos unicelulares que apresentam estratégias de alimentação semelhantes, mas não estão diretamente relacionadas ao Orciraptor. Atualmente, dezenas desses organismos são cultivados e caracterizados geneticamente no Instituto de Zoologia. Tudo isso é possível graças aos recentes avanços tecnológicos no campo do sequenciamento de alto rendimento. No entanto, trabalhar com alimentadores de protoplastos também requer conhecimentos especiais no manuseio de microrganismos exóticos. O cientista está convencido de que é hora da biologia moderna voltar à diversidade de organismos não-modelo novamente. “Os dados de nosso estudo sobre o Orciraptor destacam quão frutíferas serão as futuras análises moleculares de protistas pouco conhecidos”, disse o Dr. Hess.
Mais informações: Jennifer V. Gerbracht et al, Comparative transcriptomics reveals the molecular toolkit used by an algivorous protist for cell wall perforation, Current Biology (2022). DOI: 10.1016/j.cub.2022.05.049










