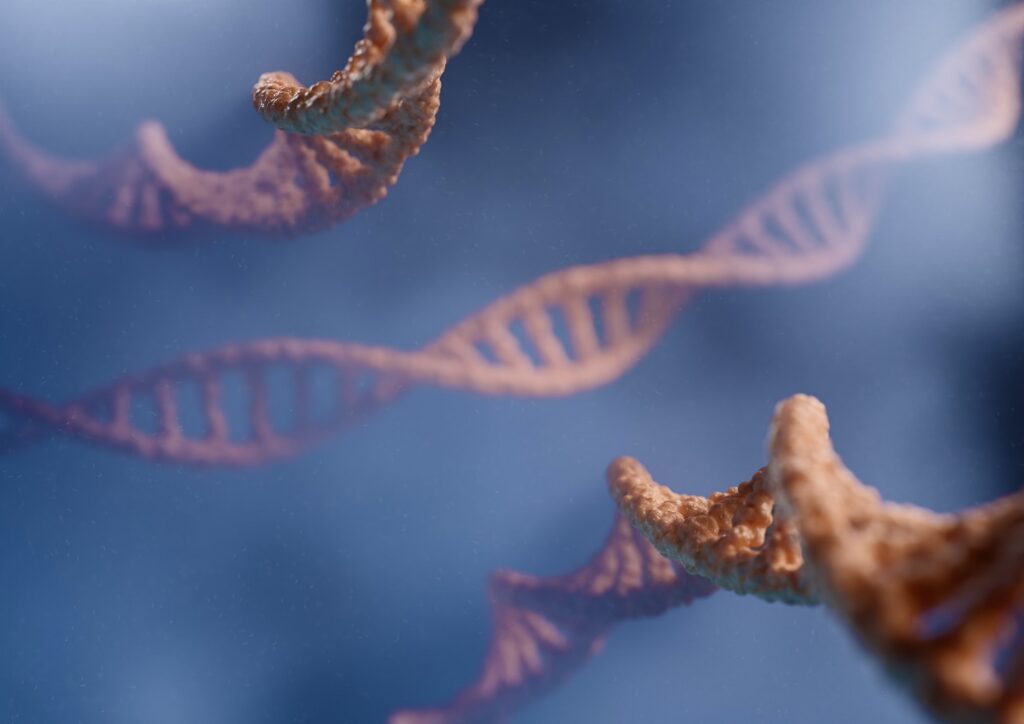
Apesar de serem densamente compactados no núcleo, os cromossomos com nossa informação genética estão em movimento, o que permite que regiões específicas entrem em contato e ativem um gene.
Por Instituto de Ciência e Tecnologia da Áustria com informações de Science Daily.
Realizar ciência de ponta requer pensar fora da caixa e reunir diferentes disciplinas científicas. Às vezes, isso significa até mesmo estar no lugar certo na hora certa. Para David Brückner, pesquisador de pós-doutorado e membro do NOMIS no ISTA, todas as coisas mencionadas acima entraram em vigor quando ele assistiu a uma palestra no campus do professor Thomas Gregor, da Universidade de Princeton. Inspirado pela palestra, Brückner apresentou uma ideia: interpretar fisicamente os conjuntos de dados específicos apresentados por Gregor. Agora, os resultados de sua colaboração são publicados na Science. Eles destacam o movimento estocástico (aleatório) de dois elementos gênicos específicos em um cromossomo, que precisam entrar em contato para que o gene se torne ativo no espaço 3D.
Como o DNA se encaixa no núcleo de uma célula
Organismos vivos como os humanos são construídos com base em genes armazenados no DNA – nosso projeto molecular. O DNA é um polímero, uma enorme molécula de partes individuais menores (monômeros). Está localizado no núcleo de cada célula.
“Dependendo do organismo, o polímero de DNA pode ter até metros de comprimento, mas o tamanho do núcleo é da ordem de mícrons”, explica Brückner. Para caber no minúsculo núcleo, o DNA é compactado ao ser enrolado como se estivesse em um carretel e ainda mais comprimido na conhecida forma dos cromossomos, que todos encontramos em um livro de biologia.
“Apesar de fortemente condensados, os cromossomos não são estáticos; eles estão se mexendo o tempo todo”, continua o físico. Essas dinâmicas são muito importantes. Sempre que um gene específico precisa ser ativado, duas regiões do polímero, chamadas de “intensificador” e “promotor”, precisam entrar em contato próximo e se ligar. Só quando isso acontece é que uma maquinaria celular lê as informações do gene e forma a molécula de RNA, que acaba dando origem a proteínas essenciais para todos os processos que um organismo vivo requer.
Dependendo do organismo, o intensificador e o promotor podem estar bem distantes um do outro no cromossomo. “Com os métodos usados anteriormente, você pode obter uma visão estática da distância entre esses elementos, mas não como o sistema evolui ao longo do tempo”, explica Brückner. Intrigados com essa informação ausente, os cientistas decidiram obter uma visão dinâmica de como esses elementos são organizados e como eles se movem no espaço 3D em tempo real.
* Continua após a propaganda.
Visualizando regiões de genes
Para atingir esse objetivo, os cientistas experimentais de Princeton estabeleceram um método para rastrear esses dois elementos de DNA durante um determinado período de tempo em um embrião de mosca. Por meio de manipulação genética, os elementos de DNA foram marcados com fluorescência, com a região intensificadora iluminada em verde e a promotora em azul. Usando imagens ao vivo (microscopia de lapso de tempo de células vivas), os cientistas foram capazes de visualizar os pontos fluorescentes em embriões de moscas para ver como eles se moviam para se encontrar.
Uma vez que os dois pontos se aproximaram, o gene foi ativado e uma luz vermelha adicional acendeu, pois o RNA também foi marcado com fluoróforos vermelhos. Brückner acrescenta entusiasmado: “Recebemos uma leitura visual de quando o intensificador e o promotor entraram em contato. Isso nos deu muitas informações sobre suas trajetórias”.
O DNA é densamente compactado e exibe movimentos rápidos
O desafio então era como analisar esse enorme conjunto de dados de movimento estocástico. Sua formação em física teórica permitiu a Brückner extrair estatísticas para entender o comportamento típico do sistema. Ele aplicou dois modelos físicos diferentes e simplificados para analisar os dados.
Um deles era o modelo Rouse. Ele assume que todo monômero do polímero é uma mola elástica. Ele prevê uma estrutura solta e difusão rápida – um movimento aleatório, onde ocasionalmente as regiões do gene se encontram. O outro modelo é chamado de “glóbulo fractal”. Prevê uma estrutura muito compacta e, portanto, difusão lenta. “Surpreendentemente, descobrimos nos dados que o sistema é descrito por uma combinação desses dois modelos – uma estrutura altamente densa que você esperaria com base no modelo de glóbulo fractal e difusão que é descrita pelas estatísticas do modelo Rouse,” Brückner explica.
Devido à combinação de empacotamento denso e movimento rápido, a ligação dessas duas regiões gênicas depende muito menos de sua distância ao longo do cromossomo do que o previsto anteriormente. “Se tal sistema está em um estado fluido e dinâmico o tempo todo, a comunicação de longa distância é muito melhor do que pensávamos”, acrescenta Brückner.
Este estudo reúne os mundos da biologia e da física. Para os físicos é interessante, porque os cientistas testaram a dinâmica de um sistema biológico complexo com teorias físicas que existem há muito tempo; e para os biólogos, fornece informações sobre as características de um cromossomo, o que pode ajudar a entender a interação gênica e a ativação gênica com mais detalhes.
Fonte da história:
Materiais fornecidos pelo Instituto de Ciência e Tecnologia da Áustria. Observação: o conteúdo pode ser editado quanto ao estilo e tamanho.
Referência do periódico :
David B. Brückner, Hongtao Chen, Lev Barinov, Benjamin Zoller, Thomas Gregor. Stochastic motion and transcriptional dynamics of pairs of distal DNA loci on a compacted chromosome. Science, 2023; 380 (6652): 1357 DOI: 10.1126/science.adf5568